Drug discovery (molecule generative model)의 성능 평가 metric들은 우리가 모델링한 어떤 generative model이 얼마나 좋은 분자들을 만들어낼 수 있는지를 평가하는 척도로, validity, novelty, uniqueness 등의 generated set 내에서 평가할 수 있는 척도들과 FCD, SA 등 test set과 generated set을 비교하여 평가할 수 있는 척도들이 있다. 이번 글에서는 다양한 molecule generative model의 성능 평가 metric들에 대해서 알아보겠다.
Implicit metrics
우선, generates set 내에서 평가할 수 있는 metric들에 대해서 알아보겠다.
Validity
Validity는 말 그대로, generate된 molecule들 중 valid한 molecule이 차지하는 비율이다. 어떤 molecule을 SMILES string 형태로 표현했을 때, 현실에서 나올 수 없는 구조를 가지는 string (CC1CCC 등)은 valid하지 못한 molecule이라고 부를 수 있다. Validity는 모델이 chemical constraint를 잘 만족하는 molecule을 만들 수 있는지를 판별하는 기준이 된다고 할 수 있다.
Uniqueness
Uniqueness는 generate된 molecule 중 중복되지 않는 unique한 molecule의 비율이다. 어떤 모델이 아무리 valid한 molecule들을 만들어낸다고 해도, 10000개의 molecule을 만들라고 했는데 항상 CCCC라는 molecule만 만들어낸다면 이는 좋은 모델이라고 하기 힘들 것이다. 그렇기 때문에 우리는 generated set의 uniqueness를 평가에 사용한다.
Novelty
Novelty는 generate된 molecule 중 training set에 포함되지 않는 molecule들의 비율이다. 우리는 training set의 분포를 잘 학습해서 새로운 molecule들을 만들어내는 것이 목적이기 때문에, training set 안에 있는 분자들만 만들어낸다면 이는 좋은 모델이라고 할 수 없을 것이다. 그렇기 때문에 우리는 novelty를 평가에 사용한다.
Internal diversity (IntDiv)
IntDiv는 generate set 내부의 checmical diversity를 판별하는 metric이다. 이는 generate set 내의 각 분자 pair들의 fingerprint 사이의 Tanimoto similarity 값을 통해 도출된다. Tanimoto similarity는 쉽게 말하면 두 비교 대상에서 겹치는 부분이 얼마나 있는지를 계산하는 metric이다. IntDiv는 다음과 같이 계산된다.
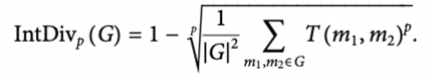
위 값은 0과 1 사이의 값으로, IntDiv 값이 클수록 더 다양한 molecule을 만들어냈다고 평가한다.
Explicit metrics (without Wasserstein distance)
다음으로, generated set을 주어진 test set과 비교하여 평가하는 metric들에 대해서 알아보겠다. Test set은 실제로 존재하는 molecule들로 구성되어 있기 때문에, 이들과 특정 property의 분포가 비슷하다면 좋은 generated set을 만들어내는 모델이라고 할 수 있을 것이다.
Fragment similarity (Frag)
Fragment similarity는 generated set과 test set 사이의 BRICS fragment의 빈도 분포를 히스토그램 형태로 비교한다. 여기서 fragment는 자주 공통적으로 도출되는 분자의 subgraph를 의미한다. 이를 계산하는 식은 다음과 같다.
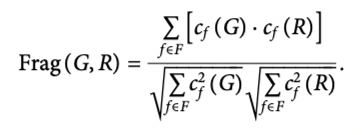
위 식에서 $c_f(G)$는 fragment f가 molecule set G에 나타나는 빈도를 말하고, R은 test set을 말한다. 위 값은 0과 1 사이의 값으로, Frag 값이 클수록 더 좋은 성능이라고 판별한다.
Scaffold similarity (Scaff)
Scaffold similarity는 fragment similarity와 비슷하게 Bemis-Murcko scaffold의 빈도 분포를 히스토그램으로 비교한다. Scaffold는 fragment와 비슷한 개념으로 분자의 subgraph를 의미한다. 이를 계산하는 식은 다음과 같다.
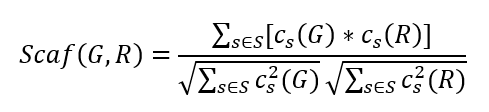
위 식에서 $c_s(G)$는 scaffold s가 molecule set G에 나타나는 빈도를 말하고, R은 test set을 말한다. 위 값은 0과 1 사이의 값으로, Frag 값이 클수록 더 좋은 성능이라고 판별한다.
Similarity to a nearest neighbor (SNN)
SNN은 각 generate된 molecule $m_G$의 fingerprint와 그것의 test set에서의 가장 가까운 neighbor $m_R$의 fingerprint 사이의 Tanimoto similarity 값의 평균을 말한다. 이를 계산하는 식은 다음과 같다.

SNN을 통해 우리는 generate된 molecule이 현실에 존재하는 다른 molecule과 얼마나 비슷하게 도출되었는지를 판별할 수 있다. 이 값 또한 클수록 좋은 성능이라고 판별한다.
Fréchet ChemNet Distance (FCD)
FCD는 generate set과 test set 사이의 biological activity 값의 분포를 비교한다. 이 biological activity 값은 분자 molecule SMILES가 ChemNet이라는 model을 통과했을 때의 prediction 값을 기반으로 한다. 이는 다음과 같이 계산된다.

이 값은 위에서 소개한 intDiv, uniqueness와 상관관계가 있다. 또한 FCD는 낮을수록 더 좋은 성능이라고 판별한다.
Explicit metrics (with Wasserstein distance)
다음으로, generated set을 주어진 test set과 비교하여 평가하는 metric들 중 Wassetstein distance를 활용하는 metric에 대해서 알아보겠다. 이도 위에서 살펴본 것과 같이 test set은 실제로 존재하는 molecule들로 구성되어 있기 때문에, 이들과 특정 property의 분포가 비슷하다면 좋은 generated set을 만들어내는 모델이라고 할 수 있을 것이다. 즉, 이 metric들은 값이 작을수록 실제 molecule의 분포를 잘 따르는 generation을 했다고 할 수 있다.
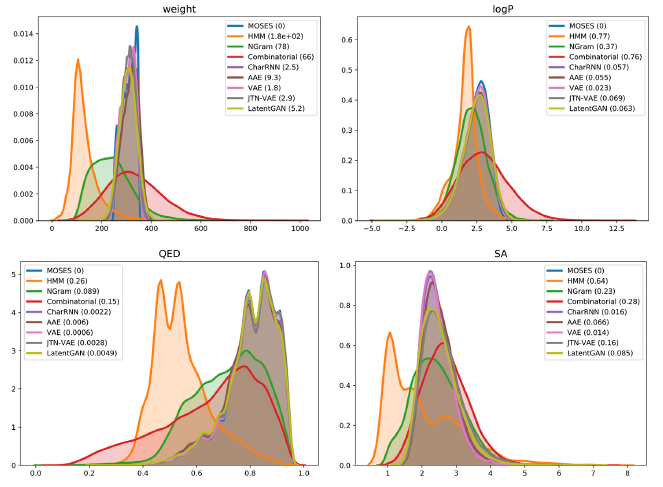
Molecule weight (MW)
MW는 generated set과 test set 간의 원자 질량 분포를 비교한다. 원자 질량을 비교함으로써 각 set이 가벼운 분자 혹은 무거운 분자에 치우쳐져 있는 것은 아닌지 비교한다.
LogP
LogP는 generated set과 test set 간의 octanol-water partition coefficient의 로그값 분포를 비교한다. Octanol-water partition coefficient는 분자가 지용성인지 수용성인지를 나타내는 지표로, 이 값에 log를 씌우면 값이 양수일 때 지용성에 가깝고, 값이 음수일 때 수용성에 가까운 특성을 가진다. 즉, generated set의 수용성/지용성 특성이 실제 데이터와 비슷한지를 비교하는 것이다.
Synthetic accessibility score (SA)
SA는 generated set과 test set 간의 합성 가능성 분포를 비교한다. 분자를 모델을 활용해 generate한다고 하더라도 실제로 합성가능한 분자를 만들어내지 않으면 실생활에서 활용할 수 있는 가능성이 낮아지기 때문에 이는 중요한 지표라고 할 수 있다. 이는 molecule 내의 fragment 조합을 기반으로 해서 측정된다.
Quantitative estimation of drug-likeness (QED)
QED는 generated set과 test set 간의 약물의 후보가 될 수 있는 가능성 분포를 비교한다. 어떤 분자가 합성이 가능하다고 하더라도 약물로 사용될 수 있는지의 여부는 이와 별개이기 때문에, 이에 대한 분석 또한 필요하다.
이번 글에서 우리는 molecular generative model의 성능을 비교하는 다양한 metric들에 대해 알아보았다. 좋은 generative model을 이해하고, 이들을 만들어내기 위해서는 우리가 최대화/최소화하기를 원하는 평가 metric들의 의미가 무엇인지를 아는 것이 필수적일 것이다.
References
Polykovskiy, D., Zhebrak, A., Sanchez-Lengeling, B., Golovanov, S., Tatanov, O., Belyaev, S., ... & Zhavoronkov, A. (2020). Molecular sets (MOSES): a benchmarking platform for molecular generation models. Frontiers in pharmacology, 11, 565644.








최근댓글