R-MAT는 relative molecule attention transformer의 준말로, 분자 내의 원자 간의 relative self-attention 정보를 반영하는 Transformer 기반의 그래프 pre-training 모델이다. R-MAT는 그래프 상 거리를 표현하는 neighborhood embedding, edge type을 반영하는 bond embedding, 그리고 radial basis function을 활용한 거리를 표현하는 distance embedding의 세 개의 embedding을 사용한다. 이번 글에서는 R-MAT가 무엇인지에 대해서 알아보겠다.
Motivation
Transformer는 absolute positional encoding, relative positional encoding 등 다양한 positional encoding을 활용한다. 그 중에서도 relative positional encoding은 각 token 사이의 상대적인 거리(relative distance)를 반영하는 역할을 한다. 예를 들어, "나는 지금 뉴올리언스에 있다"라는 문장이 있으면, "지금"과 "있다"의 거리 2를 positional encoding에 반영하는 것이다. 이러한 relative positional encoding은 다양한 task들에서 성능 향상에 기여해왔다. 기존의 molecule transformer들은 positional encoding에 대한 고려를 거의 하지 않았고, 이에 R-MAT는 원자 사이의 relative distance 정보를 활용하여 attention을 계산한다.
Method
R-MAT가 원자 사이의 relative distance 정보를 반영하는 방법은 크게 세 가지로, 1) neighborhood embedding, 2) bond embedding, 그리고 3) distanec embedding이 있다. Neighborhood embedding은 그래프 상 거리를 표현하고, bond embedding은 edge type을 반영하며, distance embedding은 radial basis function을 활용한 거리를 표현한다.
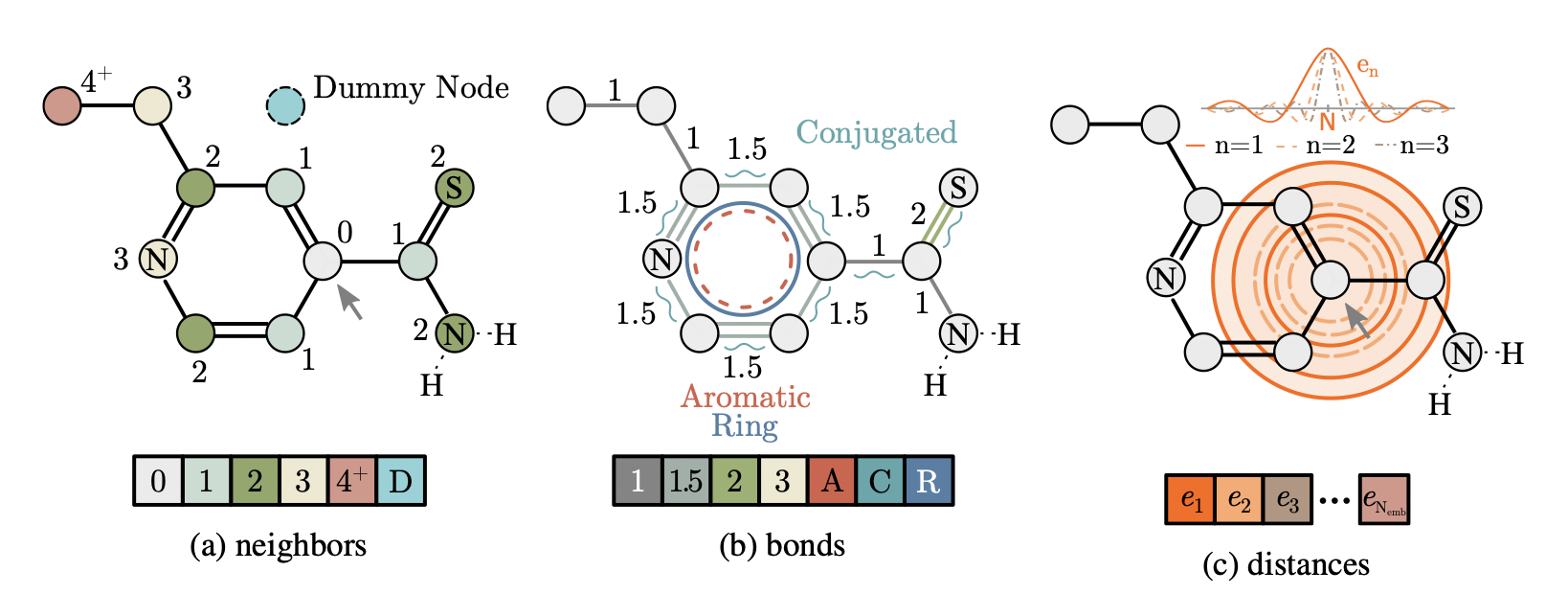
Neighborhood embedding
Neighborhood embedding은 그래프 상 거리 정보를 표현하는 embedding으로, 두 노드 사이의 shortest path 상에 얼마나 많은 노드들이 존재하는지를 의미한다. 예를 들어, 아래 그림에서 화살표가 가리키는 0으로 표시된 노드를 기준으로 옥색 노드들은 거리가 1, 초록색 노드들은 거리가 2 떨어져 있는 것이다.

Bond embedding
Bond embedding은 노드와 노드 사이에 존재하는 엣지의 엣지 타입을 의미한다. 이 엣지 타입에는 단일/이중/삼중 결합 등의 결합의 종류 뿐만 아니라 해당 엣지가 aromatic bond인지 등의 엣지의 특성들도 반영되어 있다. 이 때, 노드와 노드 사이에 결합이 존재하지 않으면 이 bond embedding은 아무런 값을 가지지 않는 0 벡터가 된다.

Distance embedding
Distance embedding은 그래프 상의 거리가 아닌 실제 3d space 상에서의 원자 사이의 거리를 의미한다. 어떤 중심이 되는 타겟 노드를 기준으로 실제 거리가 멀수록 더 큰 값을 가지는 embedding 값을 가진다.
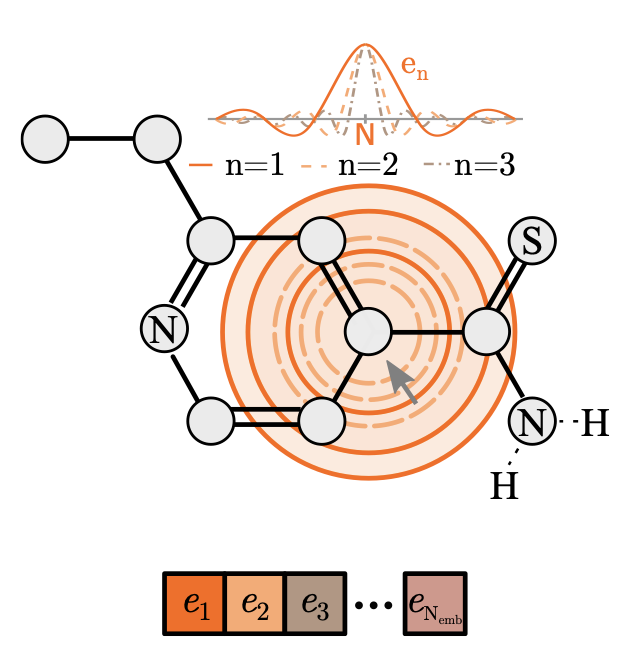
이는 두 원자 사이의 실제 거리를 기반으로 한 radial basis distance를 이용하여 계산되고, 그 식은 다음과 같다. 아래 식에서 d는 두 원자 사이의 거리, c는 cutoff distance 상수, n은 사용되는 radial basis function의 개수를 의미한다.
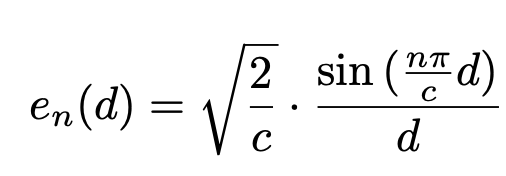
Relative molecule self-attention
위에서 설명한 relative distance 정보를 반영하는 embedding들을 활용하여 relative molecule self-attention layer를 정의할 수 있다. 우선, 우리가 위에서 얻은 embedding들을 $b_{ij}$라고 할 때, 이들을 self-attention 계산에 필요한 key와 value 값으로 매핑하기 위해 key network, value network를 정의하여 각각을 통과시킨다. 그리고 계산된 key, value 값인 $b_{ij}^K$와 $b_{ij}^V$를 이용하여 다음과 같이 query와 key의 inner product인 unnormalized attention 값에 해당하는 $e_{ij}$를 계산한다.

마지막으로, 이를 활용해 최종적인 relative molecule self-attention 값을 계산한다.

Experiments
R-MAT의 실험은 graph classification / regression task인 molecular property 예측 task에 대해서 진행되었다. 실험 결과를 통해 R-MAT가 기존의 molecular pretraining 방법들보다 더 좋은 성능을 내는 것을 알 수 있다.

이번 글에서는 R-MAT에 대해서 알아보았다. 이제 graph transformer 논문 시리즈를 마무리하고, 새로운 주제를 시작해보려고 한다.
References
Maziarka, Ł., Majchrowski, D., Danel, T., Gaiński, P., Tabor, J., Podolak, I., ... & Jastrzębski, S. (2021). Relative molecule self-attention transformer. arXiv preprint arXiv:2110.05841.








최근댓글