ESMFold는 아미노산으로 이루어진 protein sequence가 주어졌을 때 이 단백질을 구성하는 모든 원자들의 structure를 예측하는 full atomic-level structural prediction 모델로, AlphaFold가 사용했던 비효율적인 MSA 기법에 의존하지 않는 LLM 기반 모델이다. 계산이 복잡하고 비용이 많이 드는 MSA 방법을 사용하지 않음으로써, ESMFold 모델은 기존에 시간이 오래 걸리던 단백질의 구조 예측 (structure prediction)을 높은 정확도를 유지하면서 가속화하였다. 이번 글에서는 ESMFold가 무엇이고, 어떻게 작동하는지에 대해 알아보겠다.
*이 글에서 protein structure prediction은 단백질의 1차 구조(protein sequence)가 주어졌을 때, 그것의 3차 구조(tertiary structure)를 예측하는 것을 의미한다.
Motivation
전통적인 protein structure prediction은 물리 기반의 simulation을 바탕으로 이루어졌기 때문에 그 속도가 매우 느렸고, 비용도 많이 들었다. 이러한 전통적인 방법을 완전히 탈피하였던 것이 바로 AlphaFold2의 등장이다. AlphaFold2는 multiple sequence alignment (MSA)와 Transformer를 활용하여 protein sequence로부터 이의 3차원 구조(좌표)를 예측하였다. MSA는 간단히 설명하면, 서로 다른 단백질의 sequence들로부터 공통적인 부분(conserved position)이 어딘지를 찾아내고, 이러한 공통적인 부분이 공통의 조상으로부터 온 중요한 부분이라고 가정하며 패턴을 분석하는 것을 말한다.

이러한 MSA는 AlphaFold2의 핵심이 되는 구성 요소 중 하나이지만, 엄청나게 많은 양의 단백질 시퀀스를 동시에 파악하여 공통되는 부분을 찾아야 하기 때문에 계산하는 데에 비용이 많이 든다는 단점을 가진다. 이에 ESMFold 및 그것의 후속작 ESM3 모델은 이러한 MSA 과정을 패스하고, 단백질 시퀀스 자체를 글이라고 생각한다.
즉, 커다란 LLM에 엄청나게 많은 양의 단백질 시퀀스를 학습시켜 이로부터 모델이 자연스럽게 rich embedding을 얻을 수 있게 하는 것이다. 이를 통해 마치 GPT가 문법, 수학 문제 푸는 법 등을 자연스럽게 학습하듯이 protein language model은 단백질이 접히는(folding) 패턴, 구조적으로 어떤 형태가 되어야 하는지, 단백질이 어떻게 작동하는지 등을 자연스럽게 학습하는 것이다.
Architecture
ESMFold는 ESM-2, folding trunk, structure module의 세 개으 구성요소로 이루어져 있다. 한 줄로 요약하면, ESM-2는 protein sequence로부터 embedding을 학습하고, folding trunk는 이를 활용해 representation을 업데이트하며, structure module은 단백질의 3D 구조를 예측한다. 그럼 각각의 구성 요소에 대해 알아보겠다.

ESM-2
ESM-2는 sequence의 embedding을 학습하기 위해 maksed language modeling (MLM) objective를 활용한다. 즉, BERT처럼 시퀀스 내의 각 아미노산(residue)를 마스킹하여 이것이 무엇인지를 예측하는 것을 training objective로 사용하는 것이다. 이를 통해 모델은 아미노산 간의 dependency를 학습하고, 이에 따라 적절한 각 protein sequence의 representation을 학습한다.
Folding trunk
Folding trunk는 여러 개의 folding block들로 구성되어 있다.
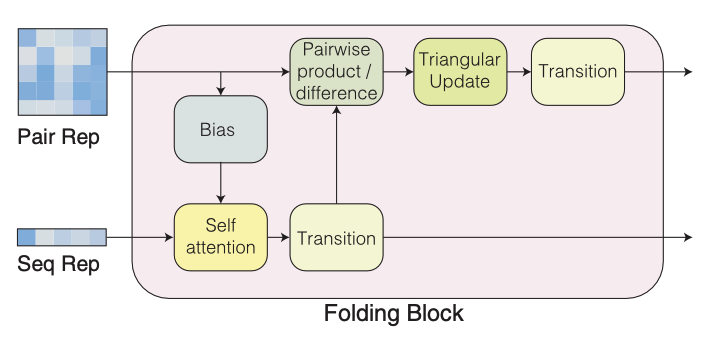
각 folding block은 단백질 시퀀스의 representation과 이 단백질 시퀀스 내의 모든 아미노산 간의 pair representation을 반복적으로 업데이트한다. 이러한 업데이트 방식은 각 아미노산의 local한 구조 정보와 각 아미노산이 pair로 서로 상호작용하는 global한 구조 정보를 모두 반영한다.
Structure module
Structure module은 앞 단계에서 업데이트한 sequence representation과 pair representation을 받아 equivariant transformer를 활용하여 단백질의 3D 구조를 예측한다. 이 때, equivariant transformer를 사용하는 이유는 단백질의 3차원 구조가 돌아가거나 이동한다면 그것이 가지는 physical property 또한 그에 맞추어서 바뀌어야 하기 때문이다.
Experiments
Structure prediction accuracy
가장 중요한 것은 이 모델이 단백질의 3차원 구조를 얼마나 잘 예측하는지일 것이다. ESMFold는 계산이 복잡한 MSA에 의존하지 않음에도 불구하고 기존의 단백질 구조 예측 모델들의 성능과 비슷한 성능을 보여주었다. 뿐만 아니라, 기존 모델들에서 MSA를 생략하였을 때와 비교하면 훨씬 좋은 성능을 보였다. 이는 MSA 없이 language model에만 의존하는 ESMFold의 구조가 잘 작동함을 증명한다.

이를 그림으로 보아도 예측한 구조와 실제 구조가 거의 일치하는 것을 확인할 수 있다.

Speed
MSA를 생략함으로써 ESMFold는 기존의 베이스라인들보다 60배 정도 빠른 inference time을 가진다. 이러한 특성은 ESMFold가 거대한 크기의 단백질 데이터셋에 대해서 효율적으로 구조 예측을 할 수 있음을 보여준다.
이번 글에서는 ESMFold가 MSA 없이 어떻게 단백질 구조 예측을 성공적으로 하였는지에 대해 알아보았다. 다음 글에서는 이의 발전된 모델인 ESM-3에 대해 알아보겠다.
아무도 안 읽을 것 같은 AI4Science 시리즈 시작! 마치 블로그 시작할 때처럼..
References
[1] Hayes, T., et al. Simulating 500 million years of evolution with a language model. Science, 2025.
[2] Baek, M., et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science, 2021.
[3] Lin, Z., et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023.
[4] Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021.
[5] Devlin, J., et al. BERT: Pre-training of deep bidirectional transformers for language understanding. NAACL-HLT, 2019.
[6] https://blog.ml6.eu/unlocking-the-secrets-of-life-ai-protein-models-demystified-f286b222d571





최근댓글