ESM-3는 단백질의 구조, 시퀀스, 그것의 기능까지 모두 고려한 multi-modal protein language model로, ESMFold에서 한 발 더 나아가 기능까지 고려한 것이 특징이다. 각 modality를 따로 학습하거나 downstream task에서만 합친 것이 아니라, pre-training 단계에서부터 이들을 통합하여 학습함으로써 서로 정보를 보완하며 학습할 수 있도록 한 것이 특징이다. 이번 글에서는 ESM-3가 무엇이고 어떻게 작동하는지에 대해 알아보겠다.
Motivation
기존의 ESMFold는 단순히 단백질의 시퀀스로부터 구조를 예측하는 하나의 태스크만을 진행할 수 있었다. 하지만 이러한 구조 예측에서 나아가, 해당 단백질이 특정 환경의 체내에서 어떤 기능을 하는지, 단백질의 구조가 기능에 어떤 영향을 미치는지 등도 단백질을 이해하는 데에 중요한 요소이고, 다양한 downstream task를 수행하는 데에 영향을 미친다. 그렇기 때문에 단백질의 시퀀스, 구조, 기능을 모두 다룰 수 있는 multi-modal 모델이 필요해졌고 이에 ESM-3가 제안되었다.

Architecture
기존 ESMFold는 masked language modeling (MLM)을 활용해서 학습을 진행해왔다. 하지만 이러한 학습 방식은 단백질이 아미노산 하나씩을 채우는 방식으로 evolve하지 않고, 구조적 기능적으로 아미노산 시퀀스를 종합적으로 고려하여 3차원 구조로 folding하는 방식으로 evolve하기 때문에 실제로 어떻게 작동하는지와는 거리가 있다. 이에 ESM-3는 시퀀스, 구조, 기능을 모두 jointly modeling하여 단백질의 특성을 좀 더 현실적으로 고려할 수 있는 학습 방식을 제시하였다.
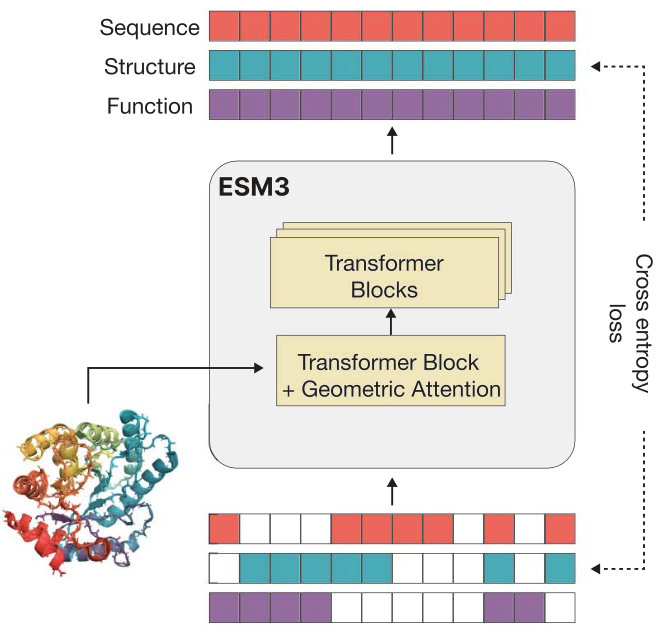
Multi-modal MLM with consistent tokenization
ESM-3는 앞서 설명했듯 single modality (sequence)의 구멍을 채우는 방식으로 학습하는 것이 아닌, 모든 modality를 고려하는 방식으로 학습을 진행한다. 기존의 방법들이 각 모달리티의 representation을 따로 학습하고 이를 마지막 단계에서 합친 것과는 다르게, ESM-3는 모든 모달리티에 대해 통합된 tokenziation 방법을 적용한다. 구체적으로, sequence는 기존과 같은 방법으로 각 residuequ별로 하나씩 tokenize하고, 3차원 구조 정보는 각 아미노산을 둘러싸고 있는 atom 정보를 discretize하며, function 정보는 각 residue의 function 정보 (catalytic site 등)를 하나씩 tokenize한다.

이러한 통일된 tokenization 구조는 각 representation이 학습 과정에서 align될 수 있게 하고, 따라서 각 모달리티가 서로의 학습에 추가적인 정보를 제공하여 더 학습이 잘 될 수 있게 도와준다.
Geometric attention
Geometric attention은 3D 좌표 정보를 더 효과적으로 처리하기 위한 장치로, 기존의 attention layer를 SE(3)-invariant하게 대체하는 것이다. 여기서 SE(3)-invariant하다는 것은 쉽게 얘기하면 돌리거나 이동시켜도 그 특성이 그대로 유지되는 것을 말한다. 구체적으로, attention은 각 residue 사이의 거리를 기반으로 하는 distance-based attention과 각 residue 사이의 상대적인 방향을 기반으로 하는 direction-based attention을 결합한다. 이러한 정보는 각 residue 사이의 관계를 효과적으로 모델링하여 최종적으로 단백질이 어떻게 folding될 것인지, 즉 3D 구조 정보가 어떻게 될 것인지를 예측하는 데에 도움을 준다.
Alignment
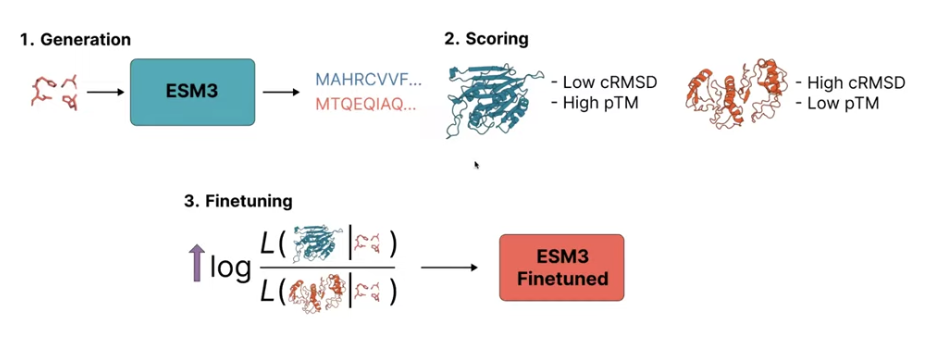
마지막으로, ESM-3는 MLM pre-training 이후에 단백질이 evolve하는 과정과 비슷한 training objective를 가지는 alignment-based fine-tuning 과정을 거친다. 이는 RLHF와 약간 비슷하게, 각 protein pair를 비교하여 더 높은 점수를 가지는 단백질을 priortize하도록 진행된다. 이 때, 이 점수는 원하는 property (cRMSD, pTM 등)로 다양하게 결정될 수 있다. 이렇게 더 높은 점수를 가지는 단백질의 likelihoood를 높이는 방식으로 학습을 진행함으로써 모델이 function에 대한 고려까지 할 수 있게 한다.
Experiments
Unconditional generation
우선, 가장 기본적인 unconditional generation 결과에 대해 살펴보겠다. 아래 그림에서 회색이 실제 단백질의 space, 색이 있는 부분이 ESM-3의 space인데, 실제에 잘 부합하도록 단백질을 고르게 생성하고 있음을 알 수 있다.

Conditional generation
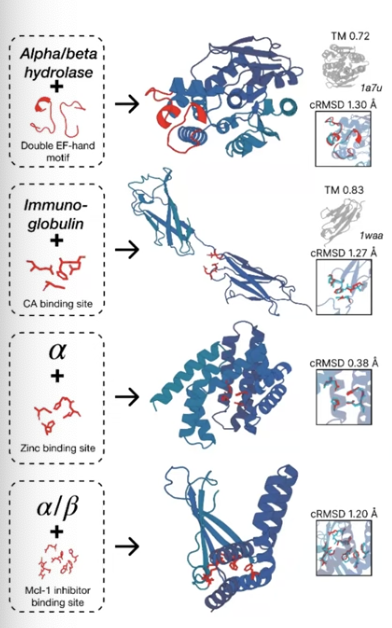
다음은 ESM-3의 진가가 가장 잘 드러나는 conditional generation이다. ESM-3는 다양한 모달리티를 모두 input으로 받을 수 있기 때문에 시퀀스가 주어졌을 때 구조를 예측하는 것 뿐만 아니라, 구조가 주어졌을 때 시퀀스를 예측하거나, 원하는 기능이 주어졌을 때 시퀀스를 예측하는 것 등 모든 것이 가능하다. 그 예시로, 아래 그림과 같이 symmetry라는 structural 정보를 조건으로 주었을 때, 아래와 같이 대칭의 단백질을 잘 생성하는 것을 확인할 수 있다.

이는 두 개 이상의 modality를 condition으로 주는 것으로 확장할 수도 있다. 예를 들어, 아래 그림에서는 structural fold와 functional site의 두 개의 모달리티를 조건으로 하는 multi-objective 단백질 structure를 예측하도록 하였는데, 이것 또한 조건에 잘 부합하도록 생성하는 것이 가능하다는 것을 확인할 수 있다.
이번 글에서는 ESM-3에 대해 알아보았다. ESM-3는 MSA 없이 language model만을 활용하여 단백질의 구조 예측을 가능하게 한 ESMFold를 확장하여 시퀀스, 구조, 기능의 모달리티를 모두 반영한 language model을 만들었고, 이를 통해 multimodal controllable generation이 가능하게 하였다. 이러한 ESM 시리즈의 발전은 LLM이 단순히 단백질의 세계를 이해하는 데에만 도움을 줄 뿐만 아니라, 과학적인 발견(scientific discovery)까지 가능하게 할 수도 있다는 것을 보인다.
References
[1] Hayes, T., et al. Simulating 500 million years of evolution with a language model. Science, 2025.
[2] Baek, M., et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science, 2021.
[3] Lin, Z., et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science, 2023.
[4] Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021.
[5] Devlin, J., et al. BERT: Pre-training of deep bidirectional transformers for language understanding. NAACL-HLT, 2019.
[6] https://blog.ml6.eu/unlocking-the-secrets-of-life-ai-protein-models-demystified-f286b222d571





최근댓글